导读
几乎所有看似无限复杂的生物学都发生在三维空间中。在生物体内,细胞必须在三维组织中相互作用和组合,每个细胞的位置与其固有性质一样重要,决定着组织的功能或疾病的功能障碍。因此,即使在研究简单生物体或单个组织的结构时,不仅需要破译成千上万个细胞的分子谱,还需要了解空间环境如何以及造成怎样的影响。近年来,随着多重空间分子检测技术的创新爆发,生物学研究开辟了新的篇章,使人们能够进一步了解生命系统的复杂性。
空间组学是继单细胞测序技术之后的又一个生物技术研究热点,其能够弥补单细胞测序技术无法获取细胞空间分布信息的缺陷。空间组学技术主要研究细胞在组织样品中的相对位置关系,用于揭示细胞空间分布关系对疾病的影响,主要检测蛋白质及其修饰或mRNA。虽然空间组学技术存在成本高、耗时长等局限,但目前这一新兴技术已为多个领域提供了大量新见解,包括动物发育和大脑结构以及与患者预后相关的肿瘤微环境(TME)的特征。
近日,英国癌症研究中心(CRUK)剑桥研究所的研究团队在Science上发表了综述文章“The dawn of spatial omics”,系统地介绍了空间组学技术的丰富种类,阐明了其原理、优势和局限性,并就这一领域目前面临的挑战提供了观点和建议。
文章发表在Science
质谱流式细胞术
在过去20年间,通过免疫组织化学(IHC)和原位杂交(ISH)技术,人们已经能够对组织进行原位检测。近年来,科研人员开发了“质谱流式细胞术(MC)”(图1),其将流式细胞技术与质谱分析技术结合在一起,使用稳定的重金属同位素(主要是镧系元素)代替荧光基团来标记抗体,并利用质谱来定量同位素标签。
MC技术可在单细胞水平同时对50种参数进行分析,包括蛋白质、核酸和小分子等,信噪比(SNR)非常高,但其目前受到足够纯金属可用性的限制。此外,该方法也是两种空间成像技术的基础,即成像质谱细胞术(IMC)和多重离子束成像(MIBI)。
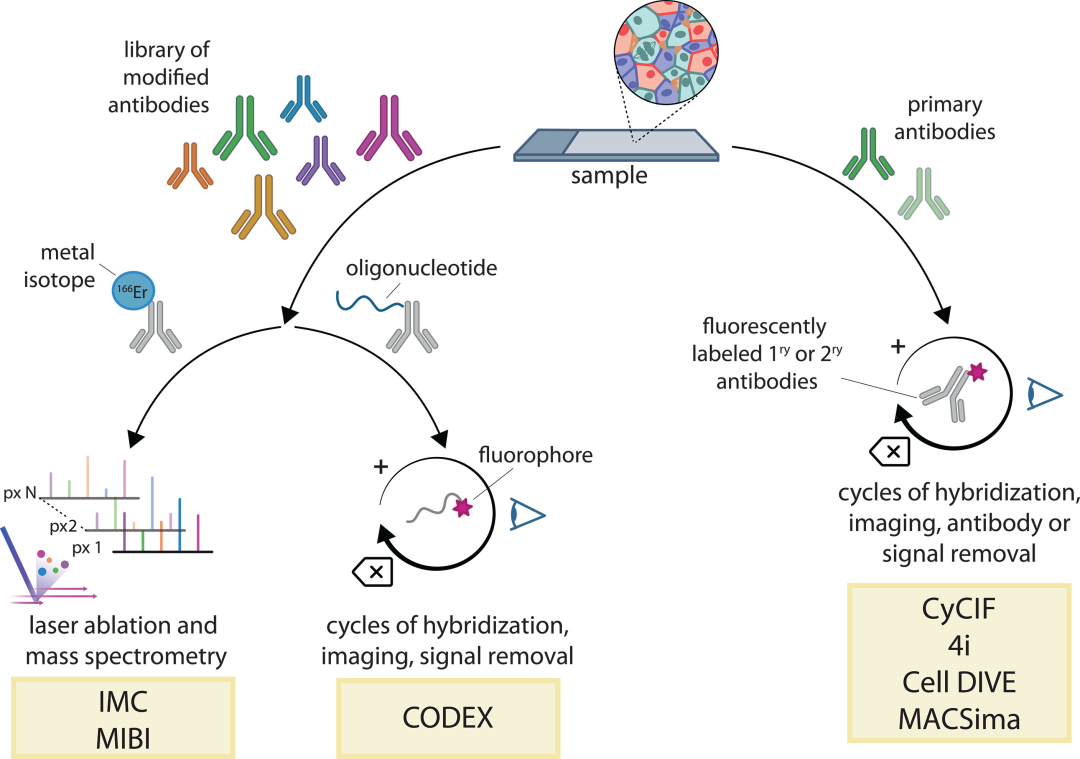
图1.基于多重抗体检测的空间蛋白质组学方法。
基于激光显微切割的转录组学
显微切割技术是指通过物理分离从特定区域纯化生物分子,即对欲选取的材料(组织,细胞群,细胞等)进行切割分离并收集用于后续研究的技术,是为分子谱分析添加空间信息的简易方法,也是空间转录组学(ST)的重要技术。显微切割能够提供非常深入的分析,几乎可达到批量测序水平,并允许科研人员将目标区域从单个细胞扩展到整个区域,但其通量相对较低。
目前,应用最广泛的显微切割技术是激光捕获显微切割(LCM)。LCM通常用于分离小区域(数十到数百个细胞)以进行RNA测序(RNA-seq),并且可以达到单细胞分辨率。LCM的优势包括广泛的转录谱、精确的组织切片以及能够与FFPE组织兼容,但难以扩展到更大数量的样本,并存在潜在的RNA降解问题。
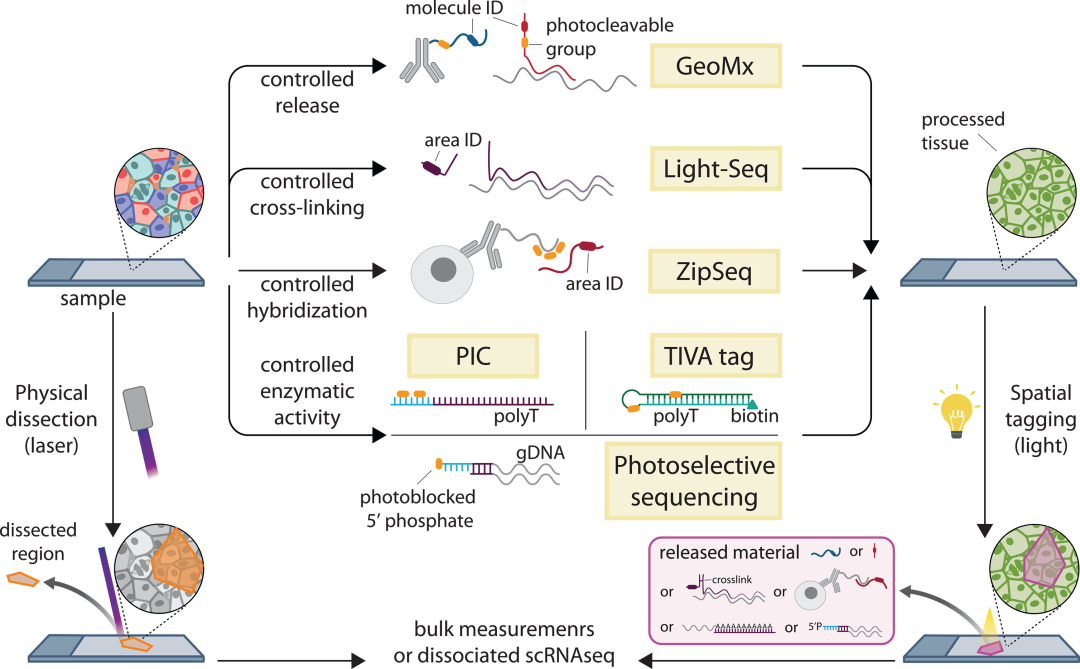
图2. 空间选择和剖析方法。
基于图像的原位转录组学
单分子荧光原位杂交(smFISH)可以同时检测多个RNA,被认为是RNA定量方法中的“金标准”。smFISH可以检测到低丰度的转录本,由此衍生的空间分析技术往往具有优异的灵敏度。循环杂交方法(osmFISH、seqFISH)是smFISH的衍生方法(图3),这是一种将组织中的单个mRNA分子分解为亚衍射荧光点的技术。为实现检测,循环杂交需要将多个探针与相同的mRNA分子进行结合,或是需要多个荧光团结合到一个小区域以产生信号,或是两个探针在几个核苷酸距离内同时结合以触发信号扩增过程。
多重容错性荧光原位杂交(MERFISH)和seqFISH+技术是目前两种主要的基于杂交的原位转录组学方法。两种方法通过结合寡聚抗体同时检测mRNA和蛋白质,可达到全转录组水平,并成功应用于不同器官和组织的空间基因表达研究。两者在灵敏度、通量和优缺点方面非常相似,但在流程操等方面存在一些技术差异。
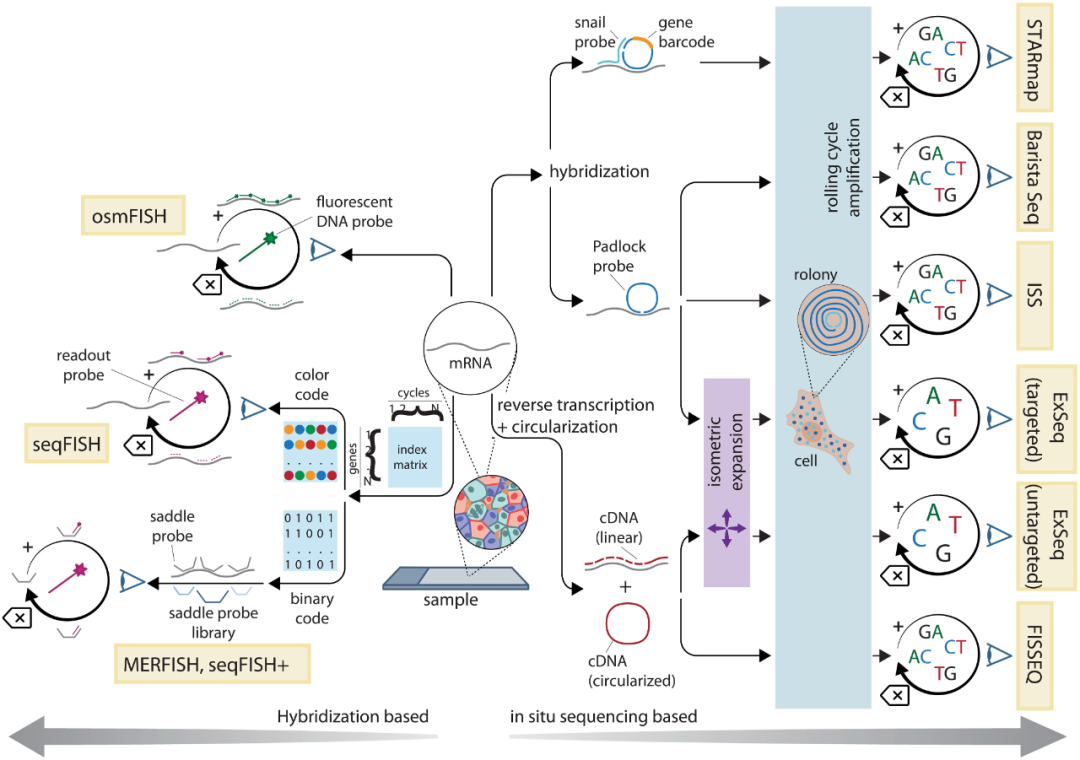
图3. 基于图像的空间转录组学方法。
基于空间条形码的转录组学
与基于图像的原位转录组技术不同,基于空间条形码(Spatial barcoding)的方法允许在整个转录组水平上对有 RNA 的物种进行无偏倚测序。空间条形码技术是通过使用空间条形码寡核苷酸阵列捕获组织RNA,将RNA序列及其空间位置关联起来(图4)。具体而言,在空间转录组学中,使用微阵列技术将有序的寡核苷酸阵列沉积在玻璃载玻片上;然后将薄的组织学切片放置在阵列上,通过渗透使细胞RNA扩散到条形码寡核苷酸;并在原位逆转录以产生空间索引的cDNA,然后将后者扩增产生文库并进行测序。
空间条形码技术的关键优势是能够产生长测序reads,不依赖于复杂的成像仪器,具有高速度以及并行化的潜力,极大地促进了商业应用。目前,空间条形码技术与批量测序相结合非常成功,但该技术仍存在一些不足,包括低分辨率(目前为50-100μm)、每个样品的成本高以及RNA捕获效率低。
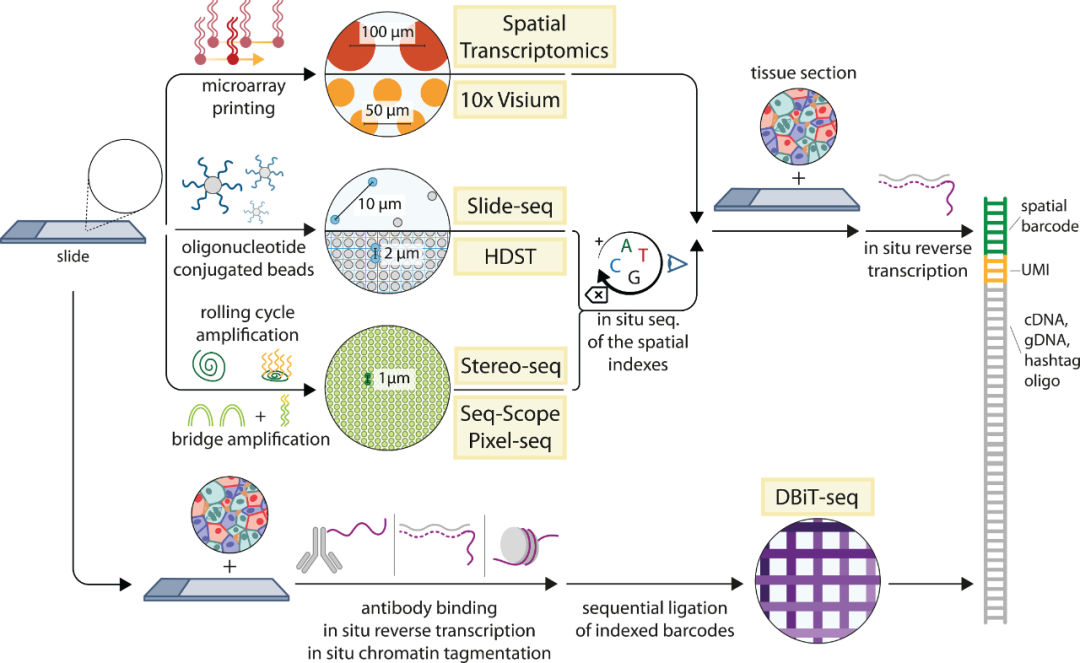
图4. 空间条形码方法。
空间组学技术面临的挑战
数据采集通常被认为是空间组学方法中最困难的步骤,此外数据操作、分析和可视化同样重要。其中,图像配准是一个非常重要的问题,巨大的数据量使空间分析变得更加复杂。目前,大多数图像配准方法采用易于识别的“基准地标”(荧光珠)作为特征来计算配准矩阵(图5),这使得配准更容易,但代价是需要额外的实验处理。
此外,大多数转录组学方法需要解码步骤,这些处理流程需要量身定制,以考虑每种技术或显微镜特有的扭曲和畸变,因此很难标准化和优化。

图5. 空间剖面数据集的分析工作流程。
展 望
空间组学领域未来的发展趋势主要体现在三个方面:1)多组学,即同时检测不同的参数(如 DNA、RNA 和蛋白质);2)增加获取和普及性,使技术变得更容易获得、更可靠、更强大;3)改进分析框架。随着技术的不断发展,需要对数据分析和实验设计给予更多关注。空间组学技术已经可以在单次实验中产生数太字节(TB)的数据,这给数据处理、分析和可视化带来了巨大挑战。
结 语
综上所述,空间组学被广泛认为是生命科学的新前沿,有望改变生物学的多个领域,并通过同时检测物理组织结构和分子特征来彻底改变病理学。虽然该领域在过去的5年里发展迅猛,但仍然面临一些挑战:进入技术壁垒,稳健性、实验设计及最佳分析实践不明确,以及缺乏标准化。该综述中,研究团队系统介绍了不同的空间组学技术,全面阐述了其优势及局限性,并给出了合理的建议。
参考文献:
Dario Bressan, Giorgia Battistoni, Gregory J. Hannon. The dawn of spatial omics. Science(2023).
DOI: 10.1126/science.abq4964
原文链接:
https://www.science.org/doi/10.1126/science.abq4964