人类DNA的主要修饰是胞嘧啶-磷酸-鸟嘌呤(CpG)中的胞嘧啶甲基化,通常称为CpG甲基化或5-mCpG。准确检测5-mCpG模式对于理解基因表达、细胞分化和印记复杂调控机制非常重要。随着长读长测序技术的进步,甲基化检测可以直接从原始序列数据中完成,从而提供了对各种修饰进行检测的可能性,而不需要对DNA进行化学处理。长读长测序最常用的方法是Oxford Nanopore Technologies(ONT)的纳米孔测序和PacBio的单分子实时(SMRT)测序。
冰岛deCODE Genetics公司的研究团队在Genome Biology发表了题为“A comparison of methods for detecting DNA methylation from long- read sequencing of human genomes”的文章,首次对长读长测序的CpG甲基化检测工具进行了系统比较,包括最新的ONT R10.4流动槽化学测序、氧化亚硫酸盐测序(oxBS)以及SMRT测序。通过分析大量基因组,研究发现纳米孔测序在7179个DNA样本中检测的CpG甲基化高度准确,与从同一抽血中分离的132个oxBS测序样本检测一致。此外,该研究引入了CpG的质量过滤器,进一步提高了纳米孔测序CpG甲基化检测的准确性,并同时过滤约30%的CpG。

文章发表在Genome Biology
主要研究内容
研究团队利用ONT的promethION流通池对7179名个体的全血样本进行了测序,同一组样本被用来研究CpG甲基化、基因表达和序列变异之间的相关性。首先,研究团队使用Nanopolish进行CpG甲基化检测,将位于彼此相距10bp以内的CpG分组,称为CpG单元。为每个CpG单元判断其对数似然比(LLR)并分类为是否“可靠”。
1 纳米孔测序和oxBS测序之间的CpG甲基化检测具有可比性
研究团队将oxBS测序的132个DNA样本作为5-mCpG率的基线,平均覆盖率为25×。通过评估oxBS数据的平均5-mCpG率和Nanopolish预测的相应平均5-mCpG率之间的Pearson相关系数来评估Nanopolish的性能,将这种相关性称为CpG平均Pearson相关系数(APC)。分析显示,两个数据集中的5-mCpG率之间的存在高度APC。
此外,通过计算每个个体的总体甲基化水平,研究团队发现纳米孔测序样本的总体甲基化水平平均低于oxBS测序样本(图1A),这些细微差异可能是由于难以将短读长序列与参考基因组精确比对,从而影响两种方法对某些CpGs的检测。
2 纳米孔测序中CpG甲基化检测的一致性
接下来,研究团队对132个个体的DNA样本进行了纳米孔和oxBS测序,并评估了皮尔逊相关性和平均绝对差(MAD),发现高覆盖率样本的相关性明显更高,MAD更低(图1B,C)。随后研究人员计算了每个样本的皮尔逊相关性,对于所有具有高序列覆盖率的CpG位点,支持CpG单元的最低纳米孔测序深度为20×,以获得其5-mCpG率的高可靠检测(图1D)。
为了捕捉甲基化预测的分布,研究团队根据oxBS测序中的甲基化率将配对数据分为四类:未甲基化、低甲基化、间甲基化和甲基化。结果表明,Nanopolish预测与oxBS检测结果一致(图1E)。将分析限制在oxBS测序中至少有25×覆盖率的CpG,在正确预测的CpG单元中,未甲基化CpG的比例最高(86%),其次是甲基化(77%),间甲基化(56%),低甲基化(52%)(图1F)。
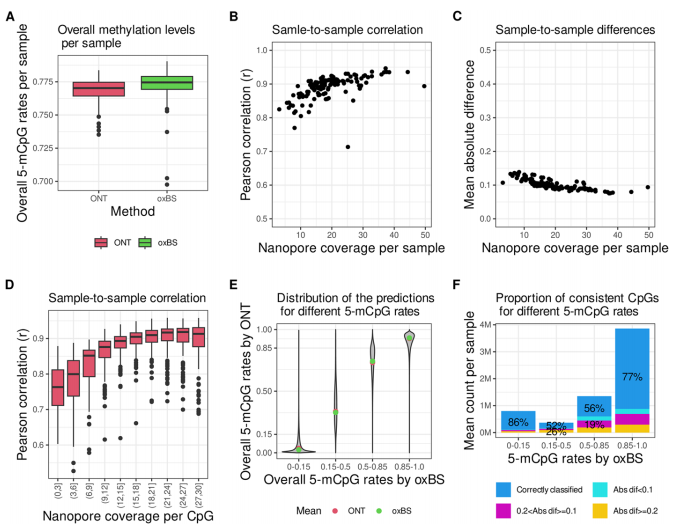
图1.纳米孔测序和oxBS测序在相同DNA样本中的性能
3 Nanopolish甲基化预测质量受CpG单元序列背景的影响
研究团队发现,与其它CpG单元相比,位于序列变异5bp内的CpG单元APC较低(图2A)。同时,oxBS测序数据中的链偏倚幅度较低,Nanopolish数据中的链偏倚较高。由于接近序列变异,从一组高质量CpG中排除了最高数量的CpG单元,其次是高链偏倚和低FRR(图2B)。值得注意的是,低甲基化(50%)和间甲基化(51%)CpG单元从一组高质量CpGs中过滤的比例高于非甲基化(17%)和甲基化(19%)(图2D)。由于高链偏倚,大多数CpGs(57.7%)从低甲基化组和间甲基化组中过滤。
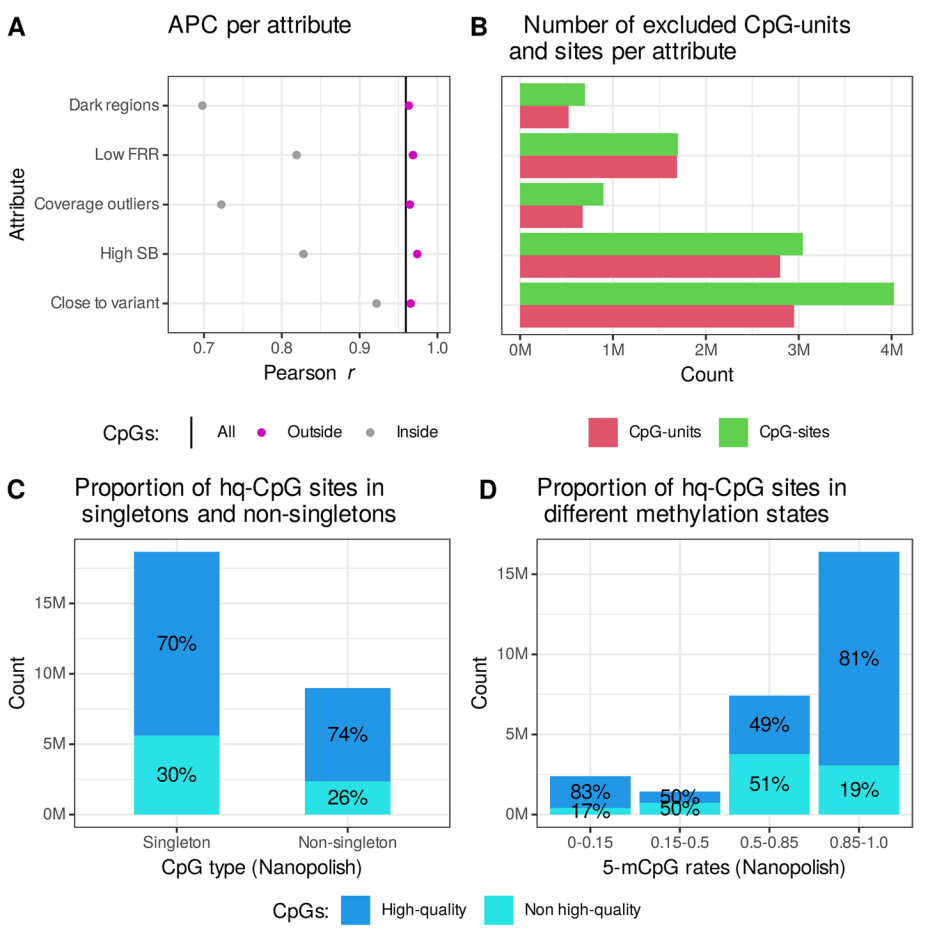
图2.通过DNA序列属性检测5-mCpG率的质量
此外,研究团队使用Guppy(版本6.2.1)预测了304个样本中CpGs的5-mCpG率。Guppy和Nanopolish的甲基化调用高度相关。Guppy的oxBS数据APC高于Nanopolish。Guppy的总体5-mCpG率相比oxBS较低。对于大多数样本,Guppy和oxBS之间的相关性高于Nanopolish和oxBS。Guppy的平均每样本链偏度和MAD也较低。
4 纳米孔测序、SMRT测序和oxBS测序的CpG甲基化检测比较
研究团队在R9.4和R10.4流动槽上对样本进行了测序。在R10.4流动槽中,所有CpGs的oxBS数据和纳米孔数据预测的5-mCpG率之间的APC更高,准确性更高。随后,研究团队对50个人的全血样本进行了SMRT测序。SMRT测序和纳米孔R9.4和R10.4测序方法的平均N50相似,但SMRT测序的平均测序错误率低于两种纳米孔测序方法中的任何一种。SMRT测序和oxBS数据中所有27,527,663个常染色体CpGs的预测5-mCpG率的APC为0.97010,MAD为0.05691。应用同样的质量过滤器后,确定了22,554,423(81.9%)个高质量CpG,APC为0.979956。
最终,研究团队比较了所有五种方法(SMRT、R9.4-Guppy、R10.4-Guppy、R9.4-Nanopolish和oxBS)之间的APC相关系数以及5-mCpG率和oxBS之间的绝对差异。应用于R10.4的Guppy和应用于R9.4的Guppy的APC最高。与oxBS相比,应用于R10.4的Guppy的APC最高,MAD最低。但方法之间观察到的APC和MAD的一些差异可能是由于样本的年龄、性别或吸烟状况的差异造成的。
5-mCpG率的分布
在50个个体的五个子集中计算所有个体的5-mCpG率,得到了所有方法的预期双峰分布(图3A,B)。相比于R9.4流动槽,Guppy应用于R10.4流动槽更接近oxBS测序样本中的甲基化分布模式。此外,所有方法显示的间甲基化CpG数量都高于oxBS测序。Guppy R10.4和SMRT的高质量CpG的分布相似,低甲基化和间甲基化CpGs的比例略低。与Nanopolish相比,Guppy R10.4和R9.4由于链偏倚和异常覆盖,过滤的CpGs较少。
功能区的5-mCpG率
研究团队计算了相对于全血中表达基因的转录起始位点(TSS)开始的50bp间隔内的平均5-mCpG率。所有甲基化检测方法都严格复制了oxBS测序样本中观察到的甲基化模式,表明TSS内缺乏甲基化(图3C)。值得注意的是,SMRT和Guppy R9.4在TSS上表现出较高的CpG甲基化率,在远离TSS的地方表现出较低的甲基化率(图3A,B)。应用于R10.4流动槽的Guppy更接近oxBS中观察到的TSS甲基化水平(图3C)。此外,Nanopolish在未甲基化的CpG单元中具有最低的MAD。
长读长测序检测到更多CpG
研究团队比较了每种基于长读长测序方法对每个样本的CpGs检测数量,发现它们都检测到相似数量的CpGs。在常染色体上,用于长读长测序的所有三种甲基化检测工具都检测了相似数量的CpGs,oxBS检测的CpGs最少(图3D)。
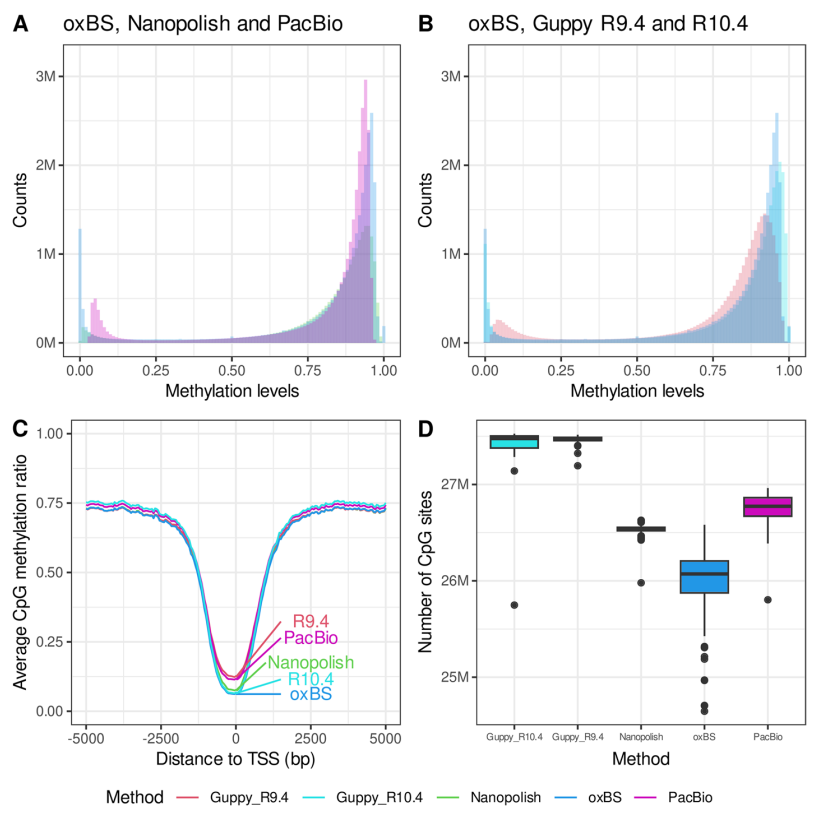
图3. CpG甲基化检测方法的比较
结 语
该研究表明基于纳米孔测序的CpG甲基化检测是高度准确的,即使对于具有高错误率的样本也是如此,SMRT测序显示了类似的结果。研究显示:
1.更高的覆盖率是CpG甲基化准确检测的重要因素;
2.纳米孔测序数据中的链偏倚在oxBS数据中未观察到。链偏倚随着较低的错误率和更准确的作图和甲基化预测而降低。
3.所有方法的甲基化预测都高度相关,并且与oxBS的5-mCpG检测一致。
4.根据研究确定的质量参数排除了CpG(过滤了7%至30%的CpG),从而提高了5-mCpG的一致性。
5.长读长测序比oxBS检测到的CpG多约3%。
论文原文:
Sigurpalsdottir, B.D., Stefansson, O.A., Holley, G. et al. A comparison of methods for detecting DNA methylation from long-read sequencing of human genomes. Genome Biol 25, 69 (2024). https://doi.org/10.1186/s13059-024-03207-9
Allergy Asthma Clin Immunol:早期宠物暴露与ADAM33甲基化模式的交互作用对过敏性鼻炎的影响
![]() 0
2021-05-22
点击查看
0
2021-05-22
点击查看

Cell Death Differ:精氨酸甲基转移酶PRMT5:基底样乳腺癌的潜在治疗靶标
![]() 0
2021-05-27
点击查看
0
2021-05-27
点击查看

Nature子刊:酒精“减”的寿,姜黄素来补救!
![]() 0
2021-10-02
点击查看
0
2021-10-02
点击查看

网络导向型SOCS3基因的循环CD4+T细胞甲基化特征与肺动脉高压患者的血流动力学之间的关系
![]() 0
2022-08-14
点击查看
0
2022-08-14
点击查看
Genome Biol:大规模食管癌甲基化综合分析揭示细胞类型和癌症特异性表观遗传调控
![]() 0
2023-09-12
点击查看
0
2023-09-12
点击查看

GRAIL发表基于甲基化模式估算ctDNA丰度的新方法TMeF,与临床分期、肿瘤大小和总生存期相关
![]() 0
2024-02-03
点击查看
0
2024-02-03
点击查看
